Por Ana Trafton
Utilizando una novedosa técnica de microscopía, investigadores del MIT y del Hospital Brigham and Women’s/Escuela de Medicina de Harvard han obtenido imágenes del tejido cerebral humano con mayor detalle que nunca, revelando células y estructuras que antes no eran visibles.
Entre sus hallazgos, los investigadores descubrieron que algunos tumores cerebrales de «bajo grado» contienen células tumorales supuestamente más agresivas de lo esperado, lo que sugiere que algunos de estos tumores pueden ser más agresivos de lo que se pensaba anteriormente.
Los investigadores esperan que esta técnica pueda eventualmente utilizarse para diagnosticar tumores, generar pronósticos más precisos y ayudar a los médicos a elegir tratamientos.
Pablo Valdés, ex postdoctorado del MIT y ahora asistente profesor de neurociencia en la Rama Médica de la Universidad de Texas es el autor principal del estudio. Por su parte, Edward Boyden, e Y. Eva Tan profesores de Neurotecnología en el MIT; e ingeniería biológica, artes y ciencias de los medios y ciencias cognitivas y del cerebro; investigador del Instituto Médico Howard Hughes; y miembro del Instituto McGovern para la Investigación del Cerebro y del Instituto Koch para la Investigación Integrativa del Cáncer del MIT; y E. Antonio Chiocca, profesor de neurocirugía en la Facultad de Medicina de Harvard y catedrático de neurocirugía en el Hospital Brigham and Women’s, son los autores principales del estudio, que aparece hoy en Science Translational Medicine .
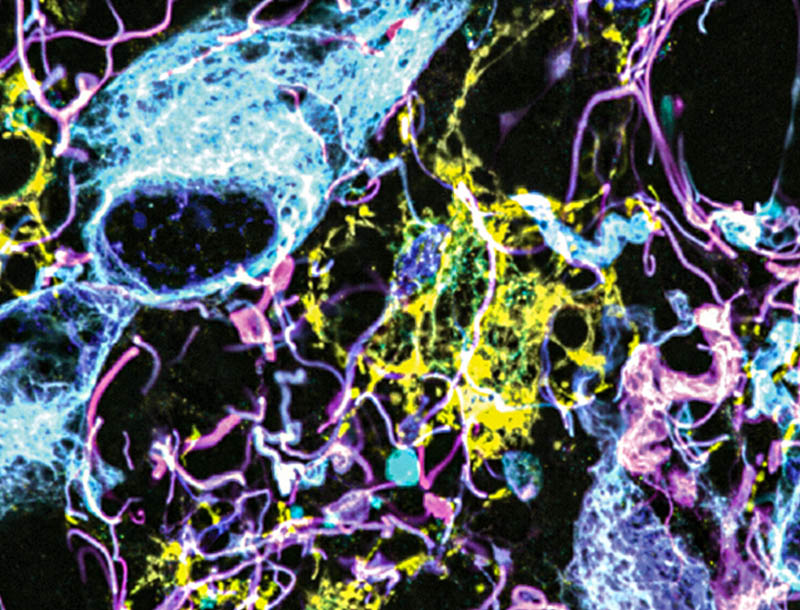
El nuevo método de obtención de imágenes se basa en la microscopía de expansión, una técnica desarrollada en el laboratorio de Boyden en 2015 basada en una premisa simple: en lugar de utilizar microscopios potentes y costosos para obtener imágenes de alta resolución, los investigadores idearon una manera de expandir el propio tejido, permitiendo se puedan obtener imágenes a muy alta resolución con un microscopio óptico normal.
La técnica funciona incrustando el tejido en un polímero que se hincha cuando se agrega agua y luego ablanda y rompe las proteínas que normalmente mantienen unido el tejido. Luego, al agregar agua, el polímero se hincha y se separan todas las proteínas. Esta ampliación del tejido permite a los investigadores obtener imágenes con una resolución de alrededor de 70 nanómetros, lo que antes sólo era posible con microscopios muy especializados y costosos, como los microscopios electrónicos de barrido.
En 2017, el laboratorio Boyden desarrolló una forma de expandir muestras de tejido humano conservadas, pero los reactivos químicos que utilizaron también destruyeron las proteínas que los investigadores estaban interesados en etiquetar. Al marcar las proteínas con anticuerpos fluorescentes antes de la expansión, se pudo visualizar la ubicación y la identidad de las proteínas una vez completado el proceso de expansión. Sin embargo, los anticuerpos que normalmente se utilizan para este tipo de etiquetado no pueden atravesar fácilmente el tejido denso antes de expandirse.
Entonces, para este estudio, los autores idearon un protocolo diferente de ablandamiento de tejidos que rompe el tejido, pero preserva las proteínas en la muestra. Una vez expandido el tejido, las proteínas se pueden marcar con anticuerpos fluorescentes disponibles comercialmente. Luego, los investigadores pueden realizar varias rondas de imágenes, con tres o cuatro proteínas diferentes marcadas en cada ronda. Este etiquetado de proteínas permite obtener imágenes de muchas más estructuras, porque una vez que el tejido se expande, los anticuerpos pueden atravesar y marcar proteínas que antes no podían alcanzar.
En colaboración con la profesora asistente del MIT, Deblina Sarkar, los investigadores demostraron una forma de esta “disminución” en 2022 utilizando tejido de ratón.
El nuevo estudio dio como resultado una técnica de despoblamiento para su uso con muestras de tejido cerebral humano que se utilizan en entornos clínicos para el diagnóstico patológico y para guiar las decisiones de tratamiento. Puede ser más difícil trabajar con estas muestras porque generalmente están incluidas en parafina y tratadas con otros químicos que deben descomponerse antes de que se pueda expandir el tejido.
En este estudio, los investigadores etiquetaron hasta 16 moléculas diferentes por muestra de tejido. Las moléculas a las que se dirigieron incluyen marcadores para una variedad de estructuras, incluidos axones y sinapsis, así como marcadores que identifican tipos de células como los astrocitos y las células que forman vasos sanguíneos. También etiquetaron moléculas relacionadas con la agresividad de los tumores y la neurodegeneración.
Utilizando este enfoque, los investigadores analizaron tejido cerebral sano, junto con muestras de pacientes con dos tipos de glioma: glioblastoma de alto grado, que es el tumor cerebral primario más agresivo y de mal pronóstico, y gliomas de bajo grado, que se consideran menos agresivo.
Para identificar células tumorales agresivas en los gliomas que estudiaron, los investigadores etiquetaron vimentina, una proteína que se encuentra en los glioblastomas altamente agresivos. Para su sorpresa, encontraron muchas más células tumorales que expresaban vimentina en gliomas de bajo grado que las que se habían observado utilizando cualquier otro método.
Cuando los pacientes con glioma se someten a cirugía, las muestras de tumores se conservan y analizan mediante tinción inmunohistoquímica, que puede revelar ciertos marcadores de agresividad, incluidos algunos de los marcadores analizados en este estudio.
Los investigadores esperan que su técnica de microscopía de expansión pueda permitir a los médicos aprender mucho más sobre los tumores de los pacientes, ayudándoles a determinar qué tan agresivo es el tumor y guiando las opciones de tratamiento. Valdés ahora planea realizar un estudio más amplio de los tipos de tumores para intentar establecer pautas de diagnóstico basadas en los rasgos del tumor que pueden revelarse mediante esta técnica.
El laboratorio de Boyden también planea utilizar esta técnica para estudiar otros aspectos de la función cerebral, en tejido sano y enfermo.